
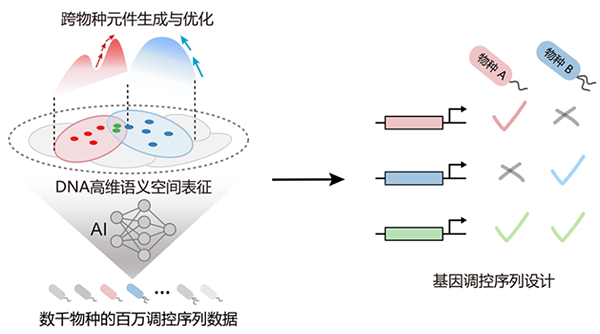
图2 基于TFBU的增强子模块化建模与智能设计
在国家自然科学基金项目(批准号:62250007、62225307)等资助下,清华大学自动化系汪小我教授团队在合成生物基因调控序列智能设计取得进展,系列研究成果连续发表两篇论文:(1)以“系统表示与优化实现细菌跨物种调控序列的逆向设计(Systematic representation and optimization enable the inverse design of cross-species regulatory sequences in bacteria)”为题,于2025年2月19日发表于《自然•通讯》(Nature Communications)期刊上。论文链接:https://doi.org/10.1038/s41467-025-57031-1;(2)以“通过引入和利用转录因子结合单元对增强子进行建模和设计(Modeling and designing enhancers by introducing and harnessing transcription factor binding units)”为题,于2025年2月8日发表于《自然•通讯》(Nature Communications)期刊上。论文链接:https://www.nature.com/articles/s41467-025-56749-2。
针对生物制造中基因线路跨宿主适配性差的瓶颈问题,研究团队从信息编码角度出发,将功能调控序列表征为DNA序列空间中的条件概率分布,发现跨物种调控规律隐含于不同物种条件概率分布的交叠区域;通过整合数千物种的百万级功能序列数据,构建了跨越物种边界的DNA高维语义表征空间和智能生成模型,突破了天然元件的物种屏障,实验结果表明:该模型在大肠杆菌和铜绿假单胞菌中实现了93.3%的跨宿主序列适配准确率(图1)。此外,针对哺乳动物细胞基因增强子定量建模难题,提出了一种新的转录因子结合单元(TFBU)模型;该模型将转录因子的核心结合位点与其周围环境序列作为一个功能整体进行建模,突破了传统方法仅关注结合位点局部组合、忽视序列上下文全局效应的局限,成功量化了环境序列对转录因子结合和增强子活性的影响,为基因治疗等新型疗法开发提供了新的工具(图2)。
系列研究将智能模型驱动的数字进化与主动学习驱动的合成生物实验相结合,通过“虚拟世界”与“物质世界”的协同探索和闭环迭代优化,显著提升了合成生物序列的设计效率。